Sulfatreduzierende Bakterien – Quantifizierung
Quantifizierung von sulfatreduzierenden Bakterien in
Wasserproben, Biofilmen oder Werkstoffen
Artikel-Nr.: 00220008
Laboranalyse: Nachweis von sulfatreduzierenden Bakterien
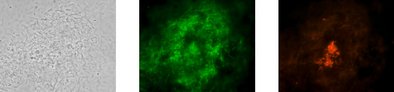
Die Proben werden auf das Vorhandensein von sulfatreduzierenden Bakterien (SRB) in Wasserproben untersucht.
Die Ergebnisse erhalten Sie bereits am 3. Werktag nach Probeneingang.
Qualitativer oder quantitativer Nachweis?
Absoluter quantitativer Nachweis
Wie erfolgt die Analyse?
Untersuchungsproben werden filtriert und unter Verwendung von spezifischen Gensonden für sulfatreduzierende Bakterien untersucht. Die Quantifizierung erfolgt als Zellzahl pro filtriertes Volumen.
Welche Anforderungen bestehen an die einzuschickende Probe?
- Wasserproben: Quantitative Analyse, mind. 150 ml
- Biofilm: Bitte fordern Sie unser Probenahmeprotokoll für Biofilmproben an.
Sie wünschen sich ein umfassendes mikrobiologisches Consulting, das speziell auf die Bedürfnisse Ihres Unternehmens zugeschnitten ist? Entdecken Sie die Möglichkeiten von VIT® Profiling
Ihre Vorteile
spezifischer Nachweis von sulfatreduzierenden Bakterien
Quantifizierung pro filtriertes Volumen
nur vitale Bakterien werden erfasst
kurze Nachweiszeit
höchste Spezifität
der Nachweis erfolgt mit der zuverlässigen VIT® Gensondentechnologie